Klinische KI-Integration
Die AG Klinische KI-Integration ist eine hybride Arbeitsgruppe zwischen den Fachbereichen Radiologie und Informationstechnologie. Sie ist entsprechend sowohl dem Institut für Diagnostische und Interventionelle Radiologie und Neuroradiologie als auch dem Institut für Künstliche Intelligenz in der Medizin (IKIM, Research group: Data Integration and AI in Radiology) angegliedert. Die Forschung fokussiert sich auf die Nutzung neuer Technologien aus der Informationstechnologie/Data Science zur Lösung bestehende Fragestellungen in der Radiologie.
Der Schwerpunkt liegt dabei auf dem Virtual Sequencing, das Machine Learning zur Generierung von Bilddaten nutzt, um z.B. durch selektive Kontrastverstärkung die benötigte Kontrastmittelmenge für die Bildgebung zu reduzieren oder durch die Generierung von Sequenzen die Akquisition von Magnetresonanztomographie-Untersuchungen zu beschleunigen, sowie auf der nicht-invasiven Tumordecodierung und der Körperzusammensetzungs- und organanalyse.
Neben der Entwicklung eigener Algorithmen in den genannten Schwerpunkten beschäftigt sich die Arbeitsgruppe mit der Integration und Validierung von KI-Anwendungen für die klinische Routine
Forschungsschwerpunkte
CT-basierte Körperzusammensetzungsanalyse als Risikofaktor
Fortschritte in der Künstlichen Intelligenz ermöglichten in den letzten Jahren rasante Entwicklungen in der vollautomatischen Quantifizierung von Geweben und Organsystemen. Mittels einer Kombination aus modernen Techniken des maschinellen Lernens und Schwellenanalyse der Röntgendichte können so auf CT-Untersuchungen des gesamten Körpers u.a. unterschiedliche Gewebetypen vollautomatisch als bildgebende Biomarker extrahiert werden. Erfolgreich konnten wir den Zusammenhang unterschiedlicher Biomarker auf Morbidität und Mortalität von akuten, wie chronischen Erkrankungen herstellen.
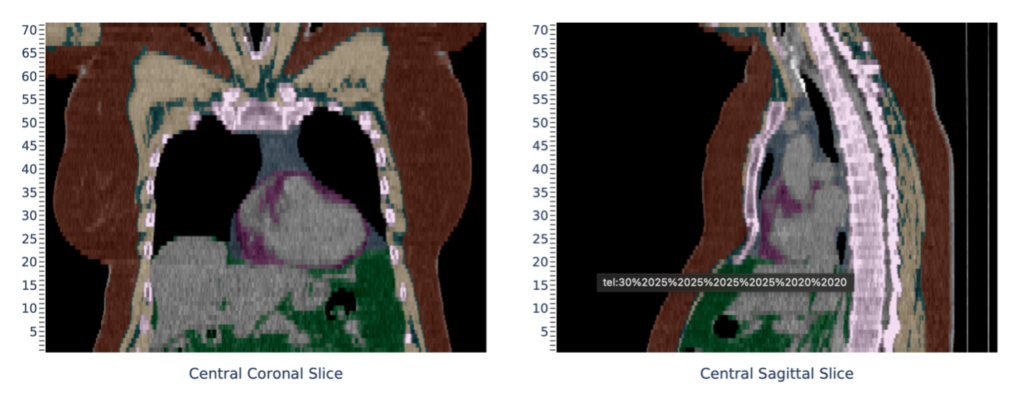
Nicht-invasive Bildgebungsgestützte Tumordecodierung
Das Universitätsklinikum Essen hat sich an einer internationalen Dosisstudie der University of California San Francisco (UCSF, USA) beteiligt, welche bereits Bei der bildgebungsgestützten Tumordecodierung werden Parameter aus den Bilddaten extrahiert, um mittels Machine Learning das genetische Profil oder das Therapieansprechen zu bestimmen sowie eine patientenindividuelle Risikostratifizierung durchzuführen. Schlüsseltechniken schließen dabei automatisierte Segmentierungsmethoden, Deep Learning oder Radiomics-basierte Featureextraktion und Machine Learning-basierte Prädiktion ein.
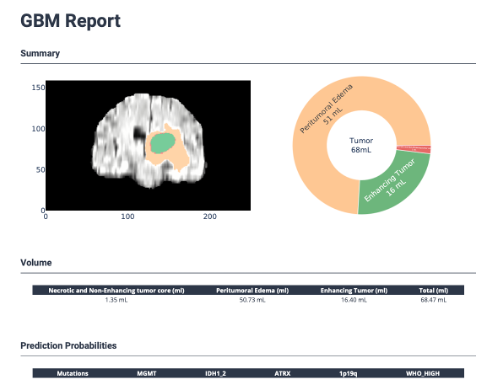
Virtual Sequencing
Beim Virtual Sequencing werden auf Deep Learning-basierend Bilddaten vorhergesagt, um bspw. MRT-Untersuchungen zu beschleunigen oder die für die Bildgebung benötigte Kontrastmittelmenge zu reduzieren. Hierzu gehört die Evaluation von Algorithmen zur Bildgenerierung wie bspw. conditional Generative Adversarial Networks, die Etablierung von Vorverarbeitungstechniken zur pixelgenauen Co-registrierung und zur Generierung von Trainingsdaten.
https://pubmed.ncbi.nlm.nih.gov/34573884/
Klinische KI-Integration
Im Rahmen der klinischen KI-Integration geht es darum, den Zugriff auf neu entwickelte Algorithmen zu erleichtern. Hierzu werden auf der einen Seite Möglichkeiten zur Integration entwickelt. Auf der anderen Seite werden fortgeschrittene Algorithmen in einem Studiensetting klinisch evaluiert und weiterentwickelt.
Kooperationen
Extern
- MR & CT Contrast Media Research (Bayer AG)
Intern
- Hybride Arbeitsgruppe (Radiologie/KI) / Teil des Instituts für künstliche Intelligenz in der Medizin (Univ.-Prof. Dr. med. Felix Nensa)
- Prof. Dr. med. Lale Umutlu
- Prof. Dr. med. Benedikt Schaarschmidt
Förderungen
2024-2027: Dr. med. Mathias Meetschen –Clinician Scientist der University Medicine Essen Clinician Scientist Academy (UMEA)
2024: Dr. med. Luca Salhöfer –Junior Clinician Scientist der University Medicine Essen Clinician Scientist Academy (UMEA)
2023: Mathias Meetschen – Junior Clinician Scientist der University Medicine Essen Clinician Scientist Academy (UMEA)
2020-2023: Dr. med. Johannes Haubold – Clinician Scientist der University Medicine Essen Clinician Scientist Academy (UMEA)
2020-2023: Kooperationspartner im Projekt “Evaluating the feasibility and accuracy of novel intra-operative molecular imaging with two PSMA-tracers in prostate cancer surgery” (DFG Sachbeihilfe – Prof. Dr. Boris Hadaschik, Prof. Dr. Ken Herrmann)
Team

Prof. Dr. med.
Johannes Haubold
Personal und KI

Dr. med.
Mathias Holtkamp, MHBA

Dr. med.
Luca Salhöfer
Data Scientist

Jannis Straus
Offene Positionen
Doktorrand (IT / Data Scientist) →
Studentische Hilfskräfte (Datenannotation) →
- Salhöfer L, Bonella F, Meetschen M, Umutlu L, Forsting M, Schaarschmidt BM, Opitz MK, Kleesiek J, Hosch R, Koitka S, Parmar V, Nensa F, Haubold J. Automated 3D-Body Composition Analysis as a Predictor of Survival in Patients With Idiopathic Pulmonary Fibrosis. J Thorac Imaging. 2024 Aug 26.
- Parmar V, Haubold J, Salhöfer L, Meetschen M, Wrede K, Glas M, Guberina M, Blau T, Bos D, Kureishi A, Hosch R, Nensa F, Forsting M, Deuschl C, Umutlu L. Fully automated MR-based virtual biopsy of primary CNS lymphomas. Neurooncol Adv. 2024 Mar 14;6(1):vdae022.
- Haubold J, Baldini G, Parmar V, Schaarschmidt BM, Koitka S, Kroll L, van Landeghem N, Umutlu L, Forsting M, Nensa F, Hosch R. BOA: A CT-Based Body and Organ Analysis for Radiologists at the Point of Care. Invest Radiol. 2024 Jun 1;59(6):433-441.
- Haubold J, Jost G, Theysohn JM, Ludwig JM, Li Y, Kleesiek J, Schaarschmidt BM, Forsting M, Nensa F, Pietsch H, Hosch R. Contrast Media Reduction in Computed Tomography With Deep Learning Using a Generative Adversarial Network in an Experimental Animal Study. Invest Radiol. 2022 Oct 1;57(10):696-703.
- Haubold J, Demircioglu A, Theysohn JM, Wetter A, Radbruch A, Dörner N, Schlosser TW, Deuschl C, Li Y, Nassenstein K, Schaarschmidt BM, Forsting M, Umutlu L, Nensa F. Generating Virtual Short Tau Inversion Recovery (STIR) Images from T1- and T2-Weighted Images Using a Conditional Generative Adversarial Network in Spine Imaging. Diagnostics (Basel). 2021 Aug 25;11(9):1542.
- Haubold, J., Hosch, R., Umutlu, L., Wetter, A., Haubold, P., Radbruch, A., Forsting, M., Nensa, F. & Koitka, S. Contrast agent dose reduction in computed tomography with deep learning using a conditional generative adversarial network. Eur Radiol 31, 6087-6095, doi:10.1007/s00330-021-07714-2 (2021).
- Haubold J, Demircioglu A, Gratz M, Glas M, Wrede K, Sure U, Antoch G, Keyvani K, Nittka M, Kannengiesser S, Gulani V, Griswold M, Herrmann K, Forsting M, Nensa F, Umutlu L. Non-invasive tumor decoding and phenotyping of cerebral gliomas utilizing multiparametric 18F-FET PET-MRI and MR Fingerprinting. Eur J Nucl Med Mol Imaging. 2020 Jun;47(6):1435-1445.